前一篇文章是2015年初开始着手,依靠自己的计算机和医学专业知识相结合完成的一篇核心期刊论文,本来这是一篇医学工程学的论文,当时还写了一个C++程序,并反复试验优化,最终三维重建的效果相当棒!但是无奈,经过杂志社反复要求,我竟然把它改成了一篇纯医学论文!自由是一件多么奢侈的事情啊~
在医学影像领域,无论是影像诊断还是影像技术,对医学图像的分析和处理都是必不可少的环节,对于人工智能在医学影像领域的发展也是重中之重。前一篇我们用到了 kitware 公司的 VTK 库,而 ITK 则是 VTK 的亲兄弟,用于图像分割和配准的开源工具包,同样支持 C++、Python 以及 Java。ITK 库实在过于专业和庞大,因此可爱的 kitware 又发布了 SimpleITK,它提供了非常易于使用的必要接口供开发人员使用,同时对 Python 的支持也更加友好。
今天这篇博文内容还算浅显,也没想好需不需要再丰富它以备正式发表,主要展示如何分割脑磁共振图像的灰质和白质。MR图像为矢状位的T1加权像,利用传统的手工方法并基于区域生长算法进行分割。ITK 需要以容积的形式将数据读入,因此我们将头MR的所有 dicom 文件放入一个文件夹,并一次性读入 ndarray,并对某个切面也就是层面进行处理和显示。
reader = SimpleITK.ImageSeriesReader()
filenamesDICOM = reader.GetGDCMSeriesFileNames(pathDicom)
reader.SetFileNames(filenamesDICOM)
img = reader.Execute()
idxSlice = 50
nda = SimpleITK.GetArrayFromImage(img)
spacing = img.GetSpacing()
figsize = (1 + margin) * nda.shape[0] / dpi, (1 + margin) * nda.shape[1] / dpi
extent = (0, nda.shape[1] * spacing[1], nda.shape[0] * spacing[0], 0)
fig = plt.figure(figsize=figsize, dpi=dpi)
ax = fig.add_axes([margin, margin, 1 - 2 * margin, 1 - 2 * margin])
plt.set_cmap("gray")
ax.imshow(nda, extent=extent, interpolation=None)
# plt.imshow(nda)
if title:
plt.title(title)
plt.show()进行下一步之前,我们需要定义两个变量,用于标识灰质和白质,以便同时显示和区分:
labelWhiteMatter = 1
labelGrayMatter = 2找到并设置区域生长的种子点的坐标:
lstSeeds = [(150, 75)]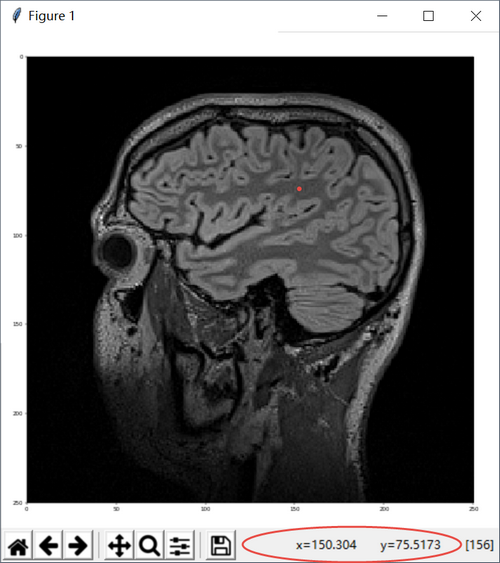
设定域值范围,开始进行区域生长,并将其标记为白质:
imgWhiteMatter = SimpleITK.ConnectedThreshold(image1=imgSmooth,
seedList=lstSeeds,
lower=130,
upper=190,
replaceValue=labelWhiteMatter)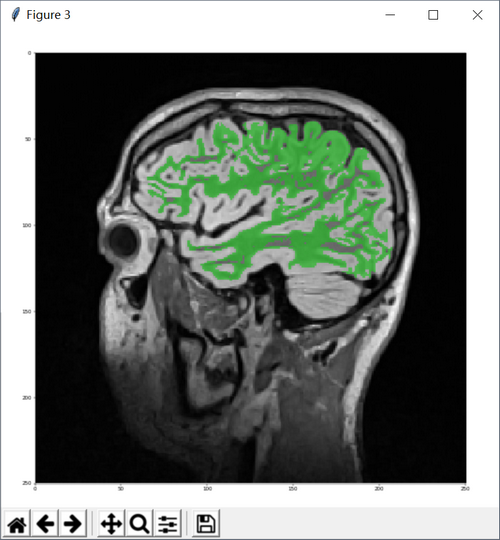
分割方法还得优化,而分割灰质的方法亦如上:
lstSeeds = [(119, 83), (198, 80), (185, 102), (164, 43)]
imgGrayMatter = SimpleITK.ConnectedThreshold(image1=imgSmooth,
seedList=lstSeeds,
lower=150,
upper=270,
replaceValue=labelGrayMatter)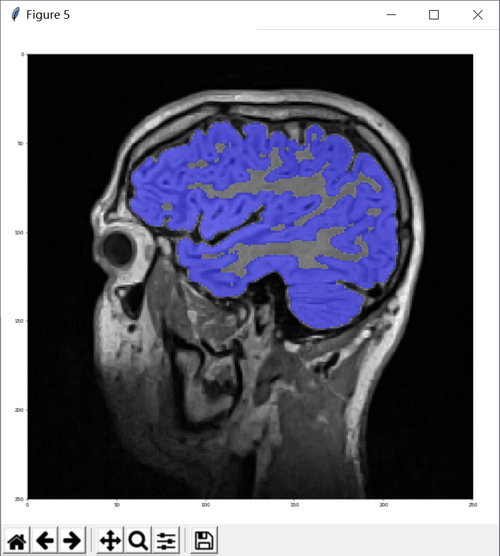
灰度图像实际上就是0-255整数分布的单通道矩阵,若要同时显示灰质和白质的分割结果,需要对矩阵进行”或”操作:
imgLabels = imgWhiteMatterNoHoles | imgGrayMatterNoHoles这样就获得了同时有两种分割标签的图像,将混合后的标签传递给 replacevalue参数,显示如下:
imgGrayAndWhiteMatter = SimpleITK.ConnectedThreshold(image1=imgSmooth,
seedList=lstSeeds,
lower=150,
upper=270,
replaceValue=imgLabels )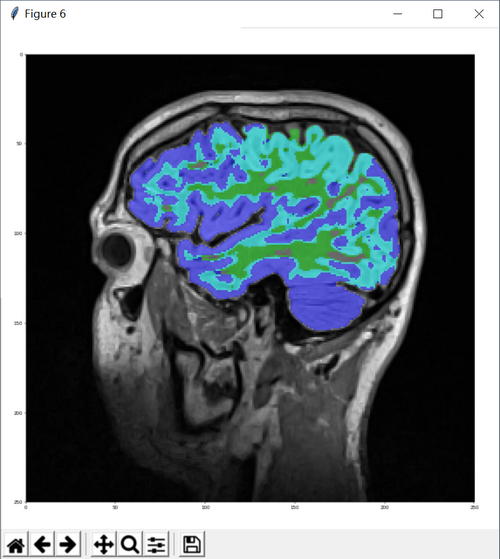
因为阈值的设定还有待优化,导致灰质的分割存在问题,进而标签混合后的结果不是特别理想,优化和微调在所难免,欢迎感兴趣的小伙伴们一起交流!
